La co-inmunoprecipitación es una de las técnicas más eficaces para investigar las interacciones proteína-proteína, especialmente cuando se trata de proteínas endógenas (ni sobreexpresadas ni marcadas). Esta técnica se usa tanto para aislar como para concentrar una proteína (proteína de interés) de una muestra que contiene miles de tipos diferentes de proteínas. La proteína de interés se aisla/concentra con un anticuerpo específico. Este anticuerpo debe poder acoplarse a un sustrato sólido (beads), lo que permite su precipitación y separación del resto de la muestra.
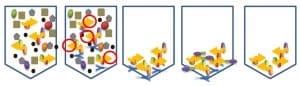
El siguiente protocolo de CO-IP usa beads de agarosa. Una alternativa a las beads de agarosa son las beads magnéticas. Este protocolo se divide en dos partes: (A) crosslinking de las proteínas con formaldehído y (B) precipitación de la proteína de interés.
El formaldehído permite la formación de puentes cruzados entre las proteínas, lo que hace posible fijar proteínas que estén interaccionando en el momento de recolectar las células. De esta forma, tras el lisado celular se mantienen unidas las proteínas que estaban interaccionando, lo que permitirá analizar estas interacciones. Sin el formaldehído durante el lisado celular las uniones entre las proteínas interactuantes se romperán y tendremos las proteínas por separado.
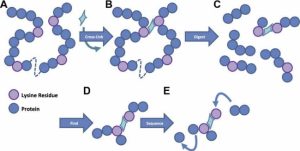
(A) CROSSLINKING:
- Sembrar las células, mantenerlas en el medio de cultivo adecuado y suplementado con el porcentaje indicado de suero, todo ello según el tipo celular. Recomendamos utilizar frascos grandes, lo ideal son T165.
- Dos días antes de que alcancen una confluencia del 95%, reducir el suero del medio de cultivo (5% de suero).
- Tras dos días creciendo con el medio de cultivo suplementado solo con un 5% de suero se recolectan células:
- Retirar el medio de cultivo.
- Lavar las células con HBSS (sin Mg+2 ni Ca+2).
- Añadir tripsina, retirar e incubar durante un minuto a 37º C.
- Recoger las células en 10 ml de medio de cultivo suplemento con 5% de suero y pasarlas a un tubo 50 ml.
- Añadir 400 µl de formaldehído e incubar 10 min a temperatura ambiente.
- Añadir 141 µl de glicina.
- Centrifugar: 5 minutos, 2000 rpm, 4ºC.
- Pasar a un tubo de 1,5 ml que ya tenga 985 µl IP-Buffer, 10 µl de PMSF y 5 μl de Cocktail de inhibidores.
- Lisar mediante movimientos de pipeta arriba y abajo y guardar a -80ºC.
(B) Co-IP:
Se necesitan dos tubos de 1,5 ml con 100 µl de beads cada uno.
Lavar las beads antes de usarlas, en ambos tubos:
- Centrifugar 100 µl de beads unos segundos, 2000 rpm, 4ºC y eliminar el sobrenadante. Las beads no se pueden centrifugar a más de 2000 rpm porque se rompen.
- Añadir 985 µl de IP-Buffer, 10 µl PMFS y 5 µl Cocktail de inhibidores.
- Centrifugar unos segundos, 2000 rpm, 4ºC. Eliminar sobrenadante.
- Repetir de tres a seis veces.
- Tras el último lavado, resuspender el pellet de beads en 100 µl IP-Buffer, 10 µl de PMSF y 5 µl de Cocktail de inhibidores.
Eliminar inespecificidades de la muestra con las beads:
- Añadir a 100 µl de beads, ya lavadas, resuspendidas en IP-Buffer, PMSF y Cocktail de inhibidores a la muestra e incubar 30 minutos a una hora, 4ºC, en agitación suave. En este paso estamos eliminando las uniones inespecíficas que se puedan formar entre los diferentes componentes de la muestra y las beads.
- Centrifugar unos segundos, 2000 rpm, 4ºC.
- Pasar el sobrenadante a otro eppendorf y tirar las beads “sucias”.
- Apartar 50 µl para otro tubo de 1,5 ml. Esto es el Whole Cell Extract (WCE) que servirá para ver el perfil proteico de la muestra antes de la Co-IP.
- Incubación del anticuerpo: Añadir a nuestra muestra, ya libre de uniones inespecíficas, el anticuerpo que reconozca específicamente la proteína de interés (optimizar concentración del anticuerpo, tener en cuenta que siempre debe estar en exceso). Incubar de una hora a dos horas a temperatura ambiente o a overnight 4ºC, en agitación suave.
- Añadir 100 µl de beads, ya lavadas, resuspendidas en IP-Buffer, PMSF y Cocktail de inhibidores a nuestra muestra que ya se ha incubado con el anticuerpo. Incubar a 4ºC overnight. Durante esta incubación el anticuerpo, que se habrá unido a la proteína de interés, se ligará a las beads, formándose el siguiente complejo:
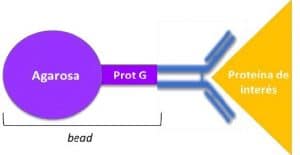
- Centrifugar unos segundos, 2000 rpm, 4ºC y desechar el sobrenadante.
- Lavar el complejo de beads, anticuerpo y proteína de interés con IP-Buffer de tres a seis veces. Para lavar, centrifugar unos segundos, 2000 rpm, 4ºC.
- Añadir al pellet de beads, anticuerpo y proteína de interés 25 µl buffer de carga (el mismo que uses para cargar las proteínas en el gel cuando haces la electroforesis) e incubar a 37ºC durante 45 minutos. Aquí estamos rompiendo la unión del anticuerpo (unido a su vez a la proteína de interés) con las beads.
- Recoger el buffer de carga para otro tubo de 1,5 ml. Añadir otros 15 µl de buffer de carga al complejo de beads, anticuerpo y proteína de interés y hervir ahora 10 minutos a 95ºC.
- Recoger el sobrenadante e incorporarlo al anterior tubo con el anticuerpo y la proteína de interés. Guardar a -80ºC.
Para comprobar la Co-IP se puede hacer un gel con tinción en plata donde se verá el perfil proteico del WCE y de la Co-IP. Como por ejemplo, el siguiente gel de plata:

En Cientisol podemos suministrar cualquier material que necesites para la realización de este tipo de aplicaciones. Si tienes cualquier consulta escríbenos a info@cientisol.com o llámanos al 981 936 338 y estaremos encantados de asesorarte.
REFERENCIAS
Autor: Gago Fuentes, Raquel. Agosto de 2022.












